Les méthodes d’analyses « omiques » - génomique, protéomique, métabolomique - génèrent aujourd’hui des quantités massives et sans cesse croissantes de données. La dissémination de l’information et de la connaissance qui en découlent ont conduit à la multiplication des ressources disponibles « en ligne » sur internet, ressources qui doivent être régulièrement mises à jour. L’une des difficultés fréquemment rencontrées est la nécessité pour un utilisateur de pouvoir étreindre d’un seul geste l’ensemble de ces ressources, ou encore de croiser les informations parfois dispersées sur toute la toile. Certains ont tenté de répondre à ce problème en créant des « repository » centralisés qui intègrent et réorganisent en leur sein des données issues d’autres ressources. L’avantage d’une telle approche est évidemment la cohérence sémantique des informations présentées ; la difficulté réside dans l’effort constant qu’il est nécessaire de fournir afin de maintenir cette cohérence et remettre à jour les informations. À l’inverse, d’autres font le choix de fédérer les ressources existantes en créant des portails capables d’interroger et d’agréger les données issues de multiples sources. Le consortium MASC-Proteomics présente un portail de cette nature : ce portail agrège un certain nombre de données expérimentales de protéomique consacrée à la plante modèle
Arabidopsis thaliana. Le portail
MASCP Gator est une page web qui permet de récupérer « à la volée » des données de protéomique à partir d’un certain nombre de ressources créées par la communauté scientifique concernée, et qui les présente sous la forme d'une interface utilisateur simple et conviviale, dont les données sont toujours à jour. Un utilisateur peut ainsi, au travers du portail, visualiser l’ensemble des informations spectrales disponibles, les modifications post traductionnelles identifiées sur les protéines, ou encore la localisation subcellulaire d’une protéine d’intérêt (
Figure). MASCP Gator apporte ainsi une aide aux scientifiques afin de concevoir de nouvelles expériences, notamment par son approche très « visuelle ». Ce type d’approche permet d’appréhender d'un coup d'œil un ensemble d’informations qui caractérisent la nature de leur protéine.
La base de données
AT_Chloro a été réalisée dans le cadre d’une collaboration entre notre laboratoire et l’équipe EDyP du
laboratoire Biologie à Grande Échelle de notre institut. Elle a été établie en réalisant près de 500 analyses par spectrométrie de masse sur des fractions hautement purifiées des compartiments intracellulaires majeurs du chloroplaste (enveloppe, stroma et thylacoïdes). AT_Chloro permet notamment de déterminer le sous-compartiment dans quel se trouve localisée une protéine du chloroplaste afin de mieux appréhender sa fonction. Cette base de données est désormais connectée au portail MASCP Gator. De ce fait, les données d’AT_Chloro peuvent être confrontées à celles contenues dans d’autres ressources. Il est par exemple possible de rechercher des protéines qui auraient été identifiés sous leur forme phosphorylée dans le cadre d’expériences réalisées par d’autres Laboratoires, suggérant ainsi une régulation probable de la fonction de ces protéines par le biais de leur niveau de phosphorylation.
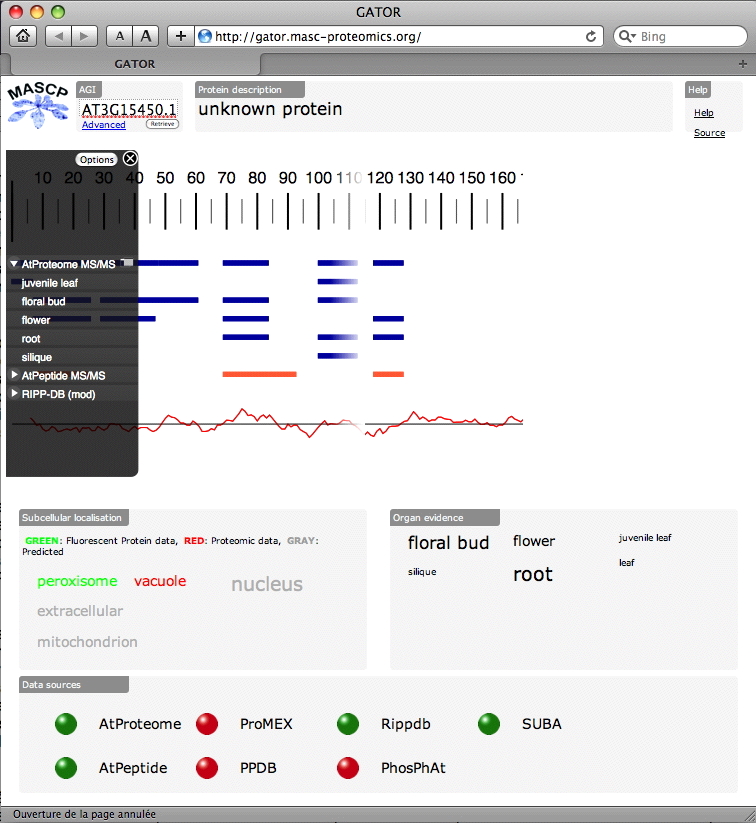
Profitant de la souplesse des technologies du web, tous les éléments constituant le portail sont disponibles sous la forme de bibliothèques « open source », pouvant être intégrées non seulement dans les ressources d'origine, mais également dans des sources secondaires telles que les wikis, ou dans des outils d’analyses ad-hoc nécessitant d’agréger des données de différentes sources.