 Responsable
Responsable
 Présentation
Présentation
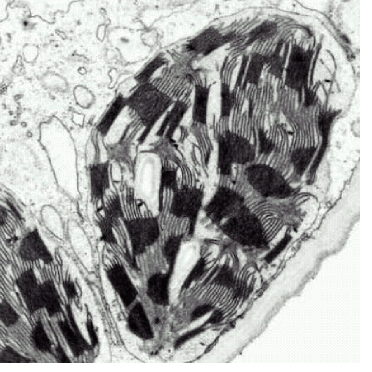 Le chloroplaste est le centre de production d'un nombre important de biomolécules (acides gras, acides aminés, sucres, vitamines, terpènes, alcaloïdes, phénols etc) qui présentent un grand intérêt pour les bioénergies comme pour l'alimentation et la santé humaine. Véritable usine énergétique de la cellule végétale, cet organite photosynthétique est responsable de la synthèse de la totalité de la biomasse de la plante, et constitue la principale « pompe à CO2 », agissant ainsi sur l'environnement et le climat. Le chloroplaste est le centre de production d'un nombre important de biomolécules (acides gras, acides aminés, sucres, vitamines, terpènes, alcaloïdes, phénols etc) qui présentent un grand intérêt pour les bioénergies comme pour l'alimentation et la santé humaine. Véritable usine énergétique de la cellule végétale, cet organite photosynthétique est responsable de la synthèse de la totalité de la biomasse de la plante, et constitue la principale « pompe à CO2 », agissant ainsi sur l'environnement et le climat. |
 Activités de recherche
Activités de recherche
Un des aspects le plus fascinant (et le moins étudié) des organismes photosynthétiques est leur capacité à gérer des variations rapides des conditions environnementales et à réorienter leur métabolisme en réponse à ce que l'on dénomme habituellement « stress » (lumière, nutriments, froid, etc…). De la rapidité de ces réponses et de leur efficacité dépend le sort de la plante dans les différentes conditions qu'elle rencontrera au cours de son cycle de vie. Ce type de réponses implique à court terme des changements métaboliques, à plus long terme des changements d'expression génique et donc de composition protéique. Notre approche vise à déchiffrer les réseaux de régulations qui contrôlent la dynamique du protéome du chloroplaste afin de mieux comprendre la capacité des plantes à répondre aux conditions variables de l'environnement.
Les approches utilisées dans l'équipe sont la protéomique à grande échelle, la microscopie confocale, des mesures de la synthèse de pigments et de vitamines, la caractérisation de transports d'ions ou de protéines. Une approche de modélisation analytique est aussi utilisée pour intégrer des données de biologie cellulaire et de biochimie. Nos organismes modèles sont les plantes supérieures (Arabidopsis, tomate, épinard…).
 Projets achevés• Grant ANR CE12 2018. Project "Polyglot". Project coordinator for the team Norbert Rolland. 2018-2023.• Grant H2020-SFS-2018-1. Projet "CSA CropBooster-P".Project participant Norbert Rolland. 2018-2022.
Projets achevés• Grant ANR CE12 2018. Project "Polyglot". Project coordinator for the team Norbert Rolland. 2018-2023.• Grant H2020-SFS-2018-1. Projet "CSA CropBooster-P".Project participant Norbert Rolland. 2018-2022.
• Grant INRA & Labex GRAL. Projet "
Chloroplast biogenesis". Coordinateur du projet Norbert Rolland. 2015-2018.
• Grant INRA. Projet "
HMA1". Coordinateurs du projet Daphné Berny/Norbert Rolland. 2014-2015.
• Grant PhD Labex GRAL & CEA. Projet "
ATPases". Coordinateur du projet Daphné Berny. 2012-2015.
• Grant Engineer Labex GRAL. Projet "
Chloroplast". Coordinateur du projet Norbert Rolland. 2012-2015.
• Grant ANR Genomics and Plant Biotechnologies. Projet "Chloro-Types". Coordinateur du projet Norbert Rolland. 2011-2014.
• Grant ANR Blanc 2010. Projet "Chloro-Pro". Coordinateur Norbert Rolland. 2011-2014.
• Grant PhD CEA IRTELIS. Coordinateur du projet Gilles Curien/Norbert Rolland. 2010-2013.
• Grant INRA. Project "Fib". Coordinateurs du projet Michel Matringe/Marcel Kuntz. 2011.
• Grant ANR Génoplante. Projet "Glyco-Chloroplast". Coordinateur de l'équipe Norbert Rolland. 2007-2010.
• Grant CNRS Post-doc. Projet “Membrane proteins”. Coordinateur du projet Norbert Rolland. 2008-2009.
• Grant CEA. Projet "CEA-PM: Screening of Alternative Expression systems for the Production of Membrane Proteins". Coordinateur du projet Norbert Rolland. 2007-2009.
• Grant PhD CEA. Projet “Atpases”. Coordinateurs du projet Daphné Berny/Norbert Rolland. 2006-2009.
• Grant CNRS Post-doc. Projet “Chlamydomonas proteomics”. Coordinateur du projet Norbert Rolland. 2005-2006.
• Grant Région Rhône-Alpes/CEA. Projet "Development of an
in vitro expression technology for the production of membrane proteins". Coordinateur du projet Norbert Rolland. 2003-2005.
• Grant Génoplante 2 - New Tools (phase 2). Projet "Characterization of
Arabidopsis thaliana gene products: Hydrophobic proteome of membranes". Coordinateur du projet Norbert Rolland. 2002 -2005.
• Grant PhD CEA. Projet “Alternative chloroplast targeting pathways”. Coordinateur du projet Norbert Rolland. 2001-2004.
• Grant Génoplante 2 - New tools. Projet "Chloroplast microarrays". Coordinateur du projet Norbert Rolland. 2001-2003.
• Grant CEA National Program of Nuclear Toxicology. Projet "Impact of radionuclides on the metabolism of higher plants". Coordinateur du projet Norbert Rolland. 2001-2003.
• Grant Génoplante I - Functional Analyses (AF 1999-035). Projet "Functional genomics of membrane transport systems in
Arabidopsis thaliana". Coordinateur de l'équipe Norbert Rolland. 2000-2001.
• Grant Génoplante I - New tools (NO 1999-3663). Projet "Characterization of
Arabidopsis thaliana gene products: Structural and functional analysis of cell and subcellular proteomes". Coordinateur de l'équipe Norbert Rolland. 2000-2001.
• Grant CEA "Action Incitative Chimie". Projet "Physiology and genetics of transport of radionuclides in plants". Coordinateur de l'équipe Norbert Rolland. 1999-2001.
• Grant CNRS “Physique et Chimie du Vivant”. Projet 1998-122 "Subcellular plant proteome: Subcellular localization of gene products of
Arabidopsis thaliana". Coordinateur du projet Norbert Rolland. 1998-2000.