LEAFY et le développement des fleurs
Article Chapeau

Publié le 12 novembre 2025
Corps de texte 1
 Fleurs et plantes à fleursDans la flore d'aujourd'hui, les plantes à fleurs ou angiospermes représentent la grande majorité des espèces végétales, mais cela n'a pas toujours été le cas. Les plantes ont commencé à se développer sur la surface de la terre voici environ 450 millions d'années. Au début, il y avait seulement des mousses, suivies plus tard par les fougères et les gymnospermes (comme le
Ginkgo biloba ou les conifères) : toutes ces plantes se reproduisent sans fleurs. Les plantes à fleurs sont apparues il y a seulement près de 130 millions d'années et elles se sont rapidement diversifiées en conquérant la plupart des écosystèmes. Grâce à l'interaction avec les insectes, les fleurs sont une structure extrêmement efficace pour la reproduction, ce qui explique le succès des angiospermes dans l'évolution.
Fleurs et plantes à fleursDans la flore d'aujourd'hui, les plantes à fleurs ou angiospermes représentent la grande majorité des espèces végétales, mais cela n'a pas toujours été le cas. Les plantes ont commencé à se développer sur la surface de la terre voici environ 450 millions d'années. Au début, il y avait seulement des mousses, suivies plus tard par les fougères et les gymnospermes (comme le
Ginkgo biloba ou les conifères) : toutes ces plantes se reproduisent sans fleurs. Les plantes à fleurs sont apparues il y a seulement près de 130 millions d'années et elles se sont rapidement diversifiées en conquérant la plupart des écosystèmes. Grâce à l'interaction avec les insectes, les fleurs sont une structure extrêmement efficace pour la reproduction, ce qui explique le succès des angiospermes dans l'évolution.
L'origine des plantes à fleurs (comment et où elles sont apparues) et de leur diversification rapide intriguait déjà Charles Darwin (qui a qualifié ce phénomène d'«abominable mystère») et est encore l'objet d'intenses études aujourd'hui. Dans notre équipe, nous étudions une protéine appelée LEAFY (LFY) qui agit comme un régulateur majeur du développement floral chez toutes les espèces angiospermes et qui était déjà présente dans les plantes sans fleurs.
En analysant cette protéine dans de nombreuses espèces de plantes, nous visons à mieux comprendre comment elle fonctionne au niveau moléculaire et comment elle a évolué et contribué à l'origine des fleurs. D'une résolution atomique aux études génomiques : études sur la structure, la fonction et l'évolution LEAFYLEAFY est un facteur de transcription (TF) original : il est présent chez toutes les plantes terrestres, il ne ressemble à aucune autre protéine et n'a pas divergé au sein d'une famille multigénique, comme l'ont fait la plupart des autres TF. Nous ne connaissons pas son origine.
D'une résolution atomique aux études génomiques : études sur la structure, la fonction et l'évolution LEAFYLEAFY est un facteur de transcription (TF) original : il est présent chez toutes les plantes terrestres, il ne ressemble à aucune autre protéine et n'a pas divergé au sein d'une famille multigénique, comme l'ont fait la plupart des autres TF. Nous ne connaissons pas son origine.
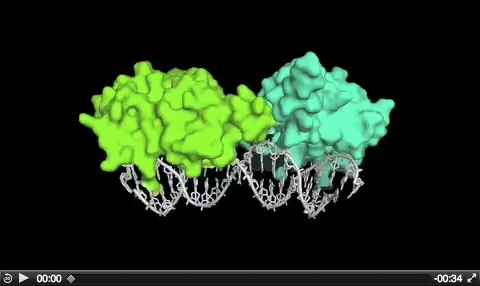
Nous utilisons des analyses biochimiques et structurales pour caractériser les propriétés de cette protéine intéressante chez de nombreuses plantes. En collaboration avec le laboratoire du Dr C. Müller, nous avons résolu la structure cristallographique du domaine de liaison à l'ADN de LFY d'Arabidopsis en complexe avec son ADN cible. Ce travail a révélé que LFY possède un motif hélice-tour-hélice au sein d'un repliement de 7 hélices encore jamais observé dans la nature. Il contacte l'ADN sur une large région de 19 paires de bases.
Film : mode de liaison de LFY à l'ADN.
 Autres vidéo de LEAFY.
Autres vidéo de LEAFY.
Images de LEAFY.
| |
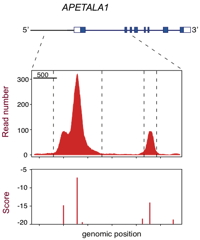
Pour comprendre comment sont ciblés les gènes que LFY régule, nous avons caractérisé et modélisé sa spécificité de liaison d'ADN (Moyroud
et al., 2011). Jusqu'à présent, on pensait que LFY liait une séquence consensus mal définie de 7-pb. Basé sur des expériences biochimiques guidées par les données structurales, nous avons construit un modèle mathématique capturant la liaison de LFY à l'ADN. Ce modèle prédit bien liaison à l'ADN au niveau génomique telle quelle se produit chez les plantes d'Arabidopsis. Nous développons plusieurs méthodes bioinformatiques pour appliquer cette méthodologie à tout facteur de transcription (Voir la page Web de
Morpheus).Légende : Le gène est régulé par APETALA1 LFY.
Les pics correspondent aux régions liées par LFY in vivo (Expérience de ChIP-seq) et les barres indiquent les sites de liaison LFY prédites par notre modèle. |
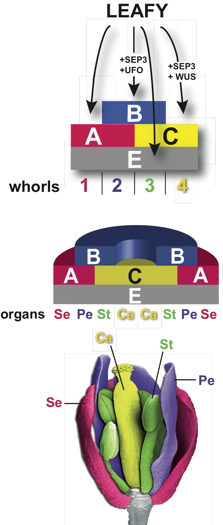
LFY bâtit la fleur en régulant des grands groupes de gènes dont les gènes homéotiques ABCE, responsables de l'identité des organes floraux. Des expériences à l'échelle du génome ont identifié des milliers d'autres gènes, liés et potentiellement régulés par LEAFY, participant à la réponse aux pathogènes, la signalisation hormonale et de d'autres voies de signalisation. Ce travail a ouvert de nombreuses nouvelles pistes pour comprendre le rôle de cette protéine fascinante.
Comme LFY est présent chez toutes les plantes terrestres (essentiellement en un seul exemplaire facilement reconnaissable), il représente une opportunité unique d'étudier l'évolution des facteurs de transcription dans le règne végétal. Nous comparons les propriétés biochimiques des protéines LEAFY de diverses plantes diverses plantes à fleurs (la vigne, le riz,
Amborella trichopoda), des gymnospermes (Ginkgo biloba,
Welwitschia mirabilis) et la mousse Physcomitrella patens. Nous essayons aussi de comprendre la fonction de LFY chez des plantes sans fleurs pour comprendre l'origine du réseau transcriptionnels qui contrôle aujourd'hui le développement des fleurs chez les angiospermes.
Légende :
LEAFY orchestre un réseau de régulations conduisant à la formation des fleurs et à l'expression de gènes homéotiques floraux. | |
 |
 Un domaine SAM d'oligomérisation façonne le paysage de liaison génomique de LEAFY
Un domaine SAM d'oligomérisation façonne le paysage de liaison génomique de LEAFY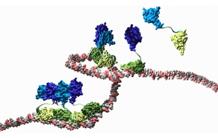 Nous avons découvert que le domaine Nterminal de LEAFY était un domaine d’oligomérisation de type SAM (Sterile Alpha Motif). Ce type de domaine est très courant dans d’autres organismes mais beaucoup moins chez les plantes. Nous avons démontré qu’il facilitait la liaison de LEAFY dans des régions dans des régions du génome qui ne possédait pas forcément des sites de très bonne affinité. De façon plus inattendue, ce domaine facilite aussi la liaison à des régions fermées du génomes suggérant qu’il confère à LEAFY des propriétés de facteur pionnier.
Nous avons découvert que le domaine Nterminal de LEAFY était un domaine d’oligomérisation de type SAM (Sterile Alpha Motif). Ce type de domaine est très courant dans d’autres organismes mais beaucoup moins chez les plantes. Nous avons démontré qu’il facilitait la liaison de LEAFY dans des régions dans des régions du génome qui ne possédait pas forcément des sites de très bonne affinité. De façon plus inattendue, ce domaine facilite aussi la liaison à des régions fermées du génomes suggérant qu’il confère à LEAFY des propriétés de facteur pionnier.
Sayou
et al.,
Nature Communications.
Lire le
communiqué de presse.
Jugé Exceptionnel sur
F1000.
| | | | |
 Publications en français
Publications en français | | |
| | | | |
| |
François Parcy
Mais qu'est-ce qui fait fleurir les plantes.
Pour la Science, 2009, n° 381, juillet 2009
Gilles Vachon, Gabrielle Tichtinsky et François Parcy
LEAFY, le régulateur clé du développement de la fleur.
Biologie Aujourd'hui, 2012,
206(1): 1-5 | | |
Haut de page