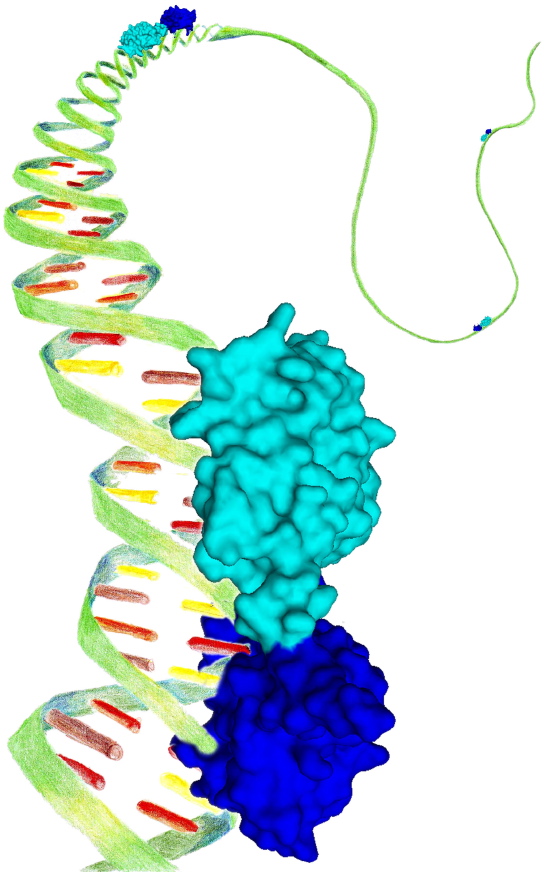
 Ces séquences d’ADN sont souvent longues de plusieurs milliers de bases et aucune méthode ne permettait jusqu’à présent d’étudier de façon quantitative la liaison des facteurs de transcription à de tels fragments. Les études se faisaient traditionnellement sur de très petites molécules d’ADN (quelques dizaines de bases). Dans cet article, nous avons, en collaboration avec le laboratoire Reproduction et Développement des Plantes de l’ENS de Lyon, montré que la technique de résonance plasmonique de surface permet de caractériser quantitativement des interactions entre un facteur de transcription et de grandes molécules d’ADN (jusqu’à 3 000 nucléotides). Pour cela, nous avons utilisé des facteurs de transcription animaux et végétaux et démontré qu’il est même possible de différencier des fragments en fonction du nombre de sites de liaison qu’ils possèdent.
Ces séquences d’ADN sont souvent longues de plusieurs milliers de bases et aucune méthode ne permettait jusqu’à présent d’étudier de façon quantitative la liaison des facteurs de transcription à de tels fragments. Les études se faisaient traditionnellement sur de très petites molécules d’ADN (quelques dizaines de bases). Dans cet article, nous avons, en collaboration avec le laboratoire Reproduction et Développement des Plantes de l’ENS de Lyon, montré que la technique de résonance plasmonique de surface permet de caractériser quantitativement des interactions entre un facteur de transcription et de grandes molécules d’ADN (jusqu’à 3 000 nucléotides). Pour cela, nous avons utilisé des facteurs de transcription animaux et végétaux et démontré qu’il est même possible de différencier des fragments en fonction du nombre de sites de liaison qu’ils possèdent.
Cette technique possède de nombreuses applications en particulier pour les études de l’évolution des réseaux transcriptionnels chez des espèces non modèles.
La résonance plasmonique de surface est une méthodes de mesure de la liaison d'un "ligand" sur un "récepteur" adsorbé à la surface d'une couche métallique. Le système de détection est basé sur une variation de l'indice de l'interface quand le ligand se fixe aux récepteurs.
Cet article a été retenu article du mois par The Plant Journal.