 IntroductionLes modifications post-traductionnelles (PTMs) affectent la structure des acides aminés composant les protéines et régulent ainsi leurs fonctions. Les PTMs sont très diverses (glycosylation, phosphorylation, acétylation, méthylation…), spontanées ou réalisées par des enzymes très spécifiques (kinases, phosphatases, méthyltransférases…), et de nature réversible ou irréversible. Les PTMs modifient tous les paramètres qui caractérisent la fonction d’une protéine, son activité, sa localisation subcellulaire, sa stabilité, ou encore l’interaction avec des partenaires protéiques ou des acides nucléiques. Toutes les grandes fonctions biologiques sont contrôlées par les PTMs (organisation de la chromatine, transcription, traduction, métabolisme, signalisation cellulaire…). L’identification et la compréhension du rôle des PTMs qui ciblent une protéine sont des éléments indispensables pour comprendre sa fonction, dans des conditions optimales de fonctionnement et sous l’effet des fluctuations de l’environnement.
IntroductionLes modifications post-traductionnelles (PTMs) affectent la structure des acides aminés composant les protéines et régulent ainsi leurs fonctions. Les PTMs sont très diverses (glycosylation, phosphorylation, acétylation, méthylation…), spontanées ou réalisées par des enzymes très spécifiques (kinases, phosphatases, méthyltransférases…), et de nature réversible ou irréversible. Les PTMs modifient tous les paramètres qui caractérisent la fonction d’une protéine, son activité, sa localisation subcellulaire, sa stabilité, ou encore l’interaction avec des partenaires protéiques ou des acides nucléiques. Toutes les grandes fonctions biologiques sont contrôlées par les PTMs (organisation de la chromatine, transcription, traduction, métabolisme, signalisation cellulaire…). L’identification et la compréhension du rôle des PTMs qui ciblent une protéine sont des éléments indispensables pour comprendre sa fonction, dans des conditions optimales de fonctionnement et sous l’effet des fluctuations de l’environnement. Activité de recherche
Activité de recherche Les recherches effectuées dans l'équipe concernent :
• la caractérisation de la voie de biosynthèse de la biotine (vitamine B8) et la biotinylation des protéines.
• l’identification de protéines méthylées ‘non-histones’ et la caractérisation du rôle de la modification post-traductionnelle dans leur fonctionnement.
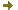 Biosynthèse de la biotine et biotinylation des protéines
Biosynthèse de la biotine et biotinylation des protéines - Nous avons identifié, caractérisé et localisé les quatre enzymes impliquées dans cette voie de synthèse. Les structures tridimensionnelles d'une de ces enzymes ont également été déterminées ; elles ont permis de comprendre le fonctionnement coordonné des deux activités portées par la protéine BIO3-BIO1 (
Figure 1). La biotine assure son rôle de transporteur de groupement carboxyle uniquement lorsqu'elle est liée de façon covalente à un nombre limité de carboxylases. Nous avons montré que cette modification post-traductionnelle très spécifique est assurée par un gène unique dont le produit est adressé vers les différents compartiments de la cellule végétale suite à une régulation de l'initiation de la traduction par un micro cadre de lecture ouverte.
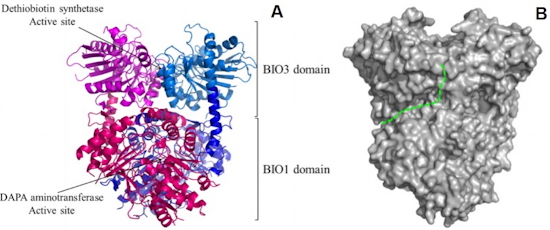 Figure 1 : Structure du dimère de l’enzyme BIO3-BIO1.
Figure 1 : Structure du dimère de l’enzyme BIO3-BIO1.
(A) Les domaines DAPA-aminotransférase (BIO1) et déthiobiotine synthétase (BIO3) de chaque monomère sont représentés en bleu foncé/clair et rouge/magenta, respectivement.
(B) La crevasse externe impliquée dans le transfert de l’intermédiaire DAPA est indiquée par la ligne verte. Identification des protéines méthylées du chloroplaste
Identification des protéines méthylées du chloroplaste - La méthylation des protéines est une modification post-traductionnelle qui contrôle la fonction biologique de nombreuses protéines cellulaires, histones et non-histones. En collaboration avec le laboratoire de Biologie à Grande Echelle (
BGE) de notre institut, nous avons édité le premier inventaire de méthylprotéines non-histones chez les plantes en utilisant comme modèle le chloroplaste d’Arabidopsis (
Figure 2). Ce méthylprotéome comprend plus d’une vingtaine de protéines modifiées sur des résidus lysine ou arginine. Elles participent au métabolisme (photosynthèse, isoprénoïdes…) et à la maintenance des chloroplastes (traduction, division…). Ces données suggèrent que la physiologie du chloroplaste peut être modulée par l’état de méthylation de certaines de ses enzymes.
 Figure 2 : Protéines méthylées dans les chloroplastes d'Arabidopsis.
Figure 2 : Protéines méthylées dans les chloroplastes d'Arabidopsis.
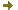 Méthylation de la protéine ribosomale L11 dans les plastes et les mitochondries
Méthylation de la protéine ribosomale L11 dans les plastes et les mitochondries - Par une combinaison d’approches nous avons montré que la protéine lysine méthyltransférase PrmA d’Arabidopsis est codée par un gène unique et adressée à la fois aux chloroplastes et aux mitochondries (
Figure 3). Dans ces deux compartiments l’enzyme assure la méthylation des protéines L11 de la grande sous-unité des plasto-ribosomes et des mito-ribosomes. Ces modifications assureraient, de concert avec d’autres PTMs décrites sur ces protéines (phosphorylation, acétylation), une régulation fine de la traduction dans les organites.
 Figure 3 : Le double adressage de la méthyltransférase PrmA dans les chloroplastes et les mitochondries conduit à la méthylation des protéines ribosomales L11 sur des résidus lysine très conservés.
Figure 3 : Le double adressage de la méthyltransférase PrmA dans les chloroplastes et les mitochondries conduit à la méthylation des protéines ribosomales L11 sur des résidus lysine très conservés.
 Décryptage de l’histoire évolutive de la méthylation de la Rubisco et des aldolases
Décryptage de l’histoire évolutive de la méthylation de la Rubisco et des aldolases - Nous avons dans un premier temps établi que la protéine lysine méthyltransférase chloroplastique LSMT, spécifique du règne végétal, est capable de méthyler deux enzymes impliquées dans l’assimilation du CO
2 au cours de la photosynthèse, la Rubisco et les fructose 1,6-bisphosphate aldolases. Chez toutes les plantes étudiées les aldolases chloroplastiques sont systématiquement modifiées alors que la méthylation de la Rubisco n’est observée que chez un nombre restreint d’espèces (Fabacées, Solanacées…). Par des approches phylogénétiques et fonctionnelles (caractérisation enzymatique de mutants et de chimères des LSMT de pois et d’Arabidopsis), nous avons décrypté les mécanismes d’évolution moléculaire qui ont conduits à la variabilité de spécificité de substrats de cette enzyme. Ainsi, seules trois mutations d’acides aminés critiques ont été nécessaires pour que la fonction ancestrale de la LSMT, la méthylation des aldolases, évolue vers la capacité de méthyler également la Rubisco (
Figure 4).
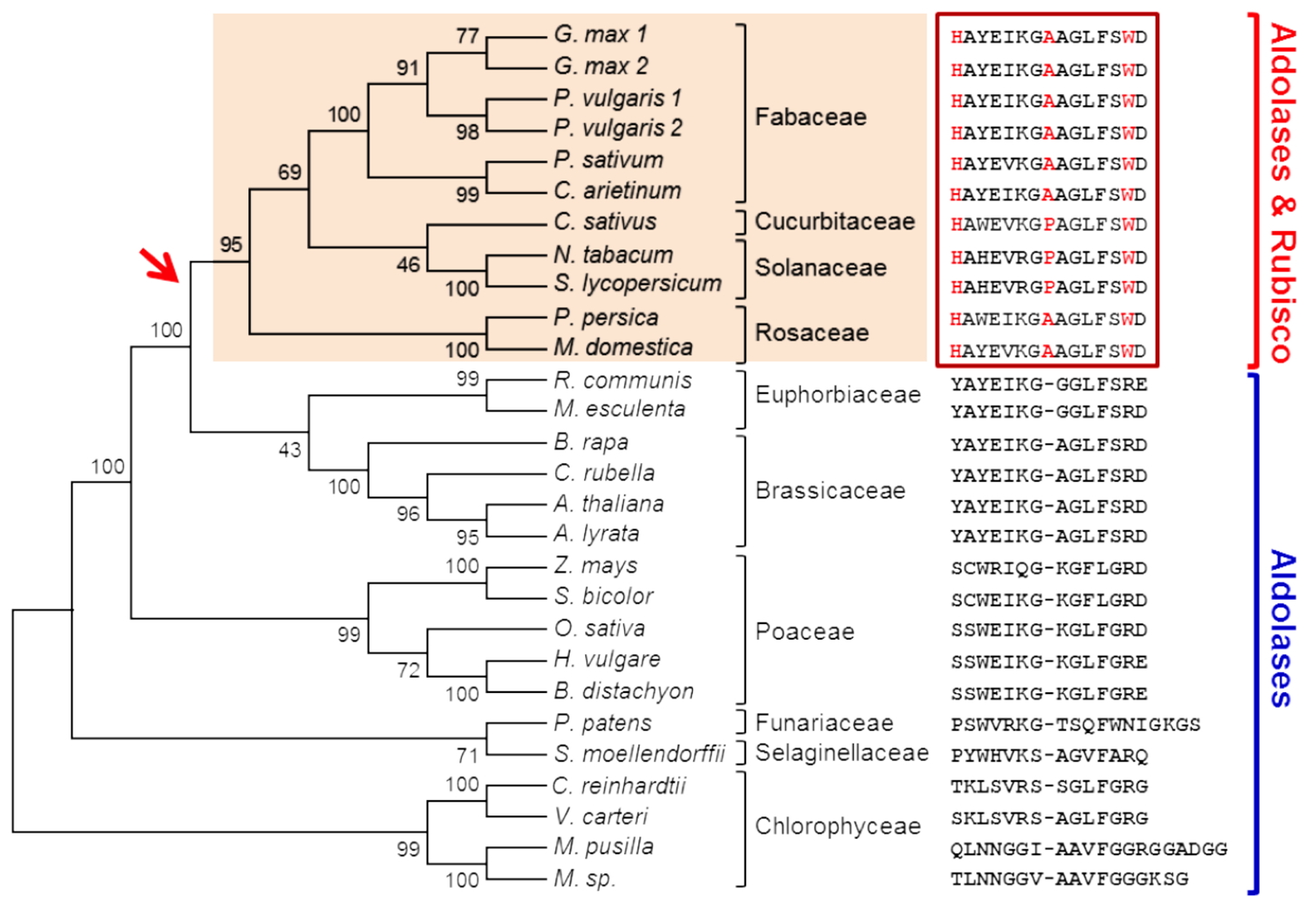 Figure 4 : Arbre phylogénétique montrant l’évolution de la spécificité de substrats des méthyltransférases LSMT.
Figure 4 : Arbre phylogénétique montrant l’évolution de la spécificité de substrats des méthyltransférases LSMT. Mots-clefs
Mots-clefs
Biotine, biosynthèse, biotinylation, méthylation, protéines méthylées, chloroplaste, mitochondrie, évolution.