Si le métabolisme cellulaire est extrêmement complexe, en particulier chez les plantes, il apparait désormais possible d’envisager une compréhension intégrée de celui-ci et d’en prédire le comportement. Ceci ne peut cependant se faire que grâce à des logiciels adaptés issus de différentes approches de modélisation.
C’est à cette tâche de modélisation que nous nous sommes attelés avec des chercheurs du laboratoire Biologie à Grande Echelle. Ceci a conduit au développement de
ChloroKB, une application Web dédiée à la plante modèle
Arabidopsis thaliana et dont l’originalité majeure réside dans la représentation graphique du contexte biologique (localisation subcellulaire des protéines et des métabolites, complexes protéiques, régulations fines).
A l’issue d’un travail de cinq ans, mobilisant biologistes et bioinformaticiens, il est désormais possible, grâce à
ChloroKB, d’explorer de manière interactive le réseau métabolique du chloroplaste et de ses inter-connexions. Ce réseau a été reconstruit manuellement en intégrant plus de 1100 protéines, 1500 métabolites et 700 complexes, localisés dans 5 compartiments subcellulaires.
ChloroKB génère des cartes graphiques basées sur le standard de modélisation « CellDesigner ». Il est possible de naviguer de manière dynamique au sein de ces cartes (au format vectoriel) qui représentent les voies métaboliques ainsi que les processus d’assemblage et de régulation des protéines impliquées dans ces voies. La prise en compte du contexte biologique, l’intégration de données hétérogènes expertisées et l’organisation hiérarchique des processus biologiques font de
ChloroKB un outil visuel analytique très original dans le paysage actuel des bases de données métaboliques. La publication de
ChloroKB constitue une ressource essentielle pour une modélisation quantitative et prédictive du métabolisme des plantes.
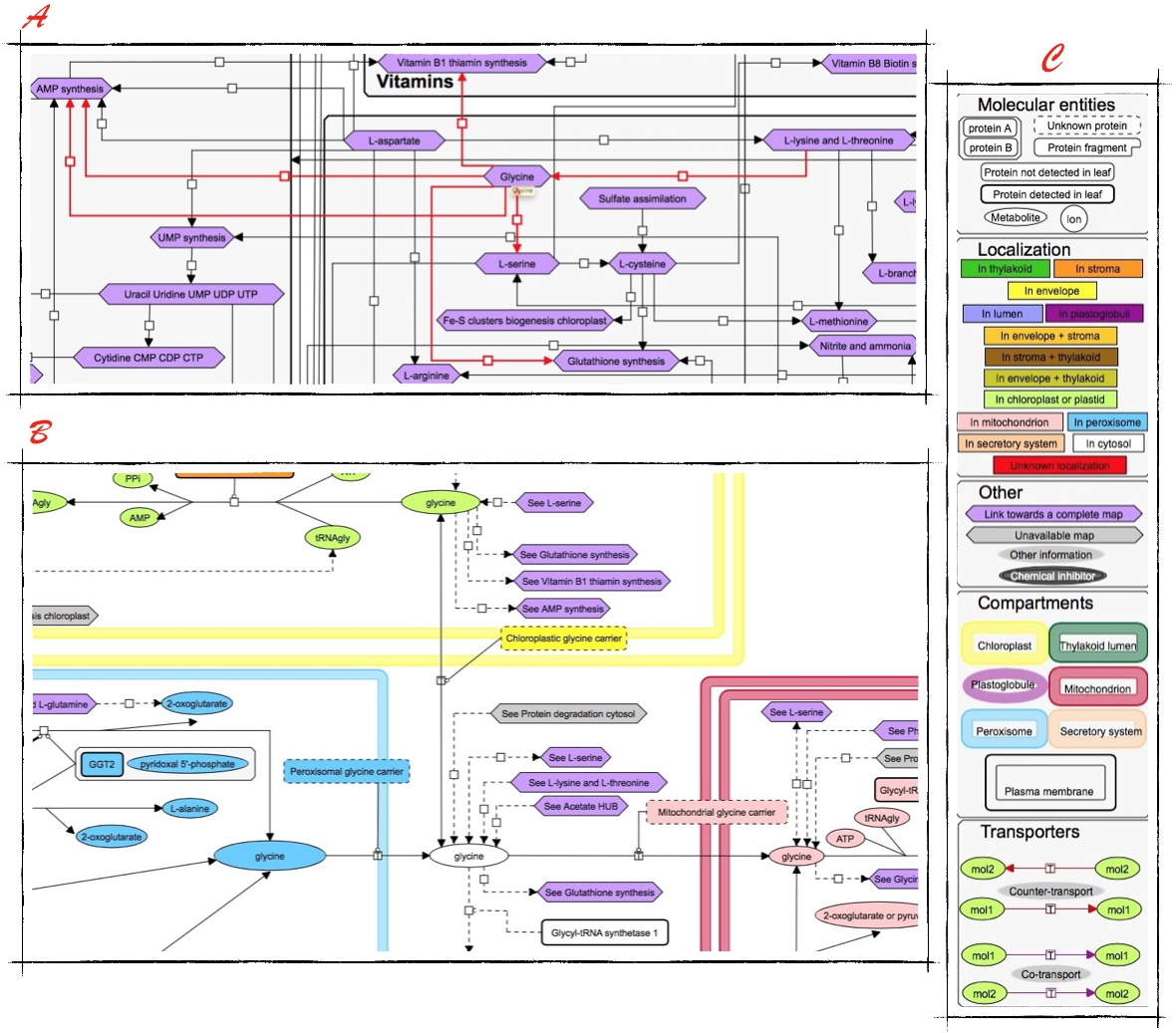
ChloroKB.
A - Extrait et zoom de ChloroKB avec la Glycine prise comme exemple. B - Extrait d’une carte métabolique. C - Légende donnant une indication de la richesse de ChloroKB.
Ce travail a été financé par le Labex GRAL.